Nucleosoma
El nucleosoma es una estructura que constituye la unidad fundamental de la cromatina, que es la forma de organización del ADN en las células eucariotas, es decir, la unidad básica de repetición de la cromatina eucariótica. Un nucleosoma se compone de alrededor de 150 pares de bases de ADN enrolladas alrededor de un núcleo de histonas. Los nucleosomas se organizan como cuentas de un collar las cuales, a su vez, son plegadas sobre sí mismas repetidas veces para formar un cromosoma.[1]
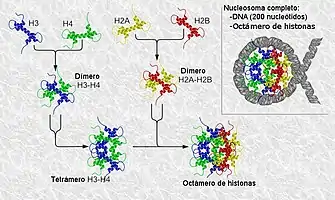
Los nucleosomas están formados por un octámero de proteínas histonas y aproximadamente 146 pares de bases nitrogenadas de ADN. [2]
El octámero está formado por dos moléculas de cada una de las histonas H2A, H2b, H3 y H4.
Las histonas son proteínas ricas en aminoácidos básicos y muy conservadas a través de la escala filogenética.[3]
Descubrimiento
En 1974, los investigadores Don y Ada Ollins fueron los primeros en observar los nucleosomas, mediante microscopía electrónica, como partículas esféricas de 70 Å de diámetro en la cromatina.[4] En el mismo año, Roger Kornberg los identificó y propuso su estructura, basándose en las siguientes en los siguientes resultados:[5]
- La cromatina contiene aproximadamente el mismo número de moléculas de histonas H2A, H2B, H3 y H4 y no más de la mitad de H1.
- La H1 reside fuera de la partícula nuclear del nucleosoma, esta se une al ADN espaciador que conecta una partícula del nucleosoma con la siguiente partícula.
- La cristalografía de rayos X indicó que en la cromatina existe una estructura regular que se repite cada 100 Å sobre la fibra. Este mismo patrón se observa cuando ADN purificado se mezcla con cantidades equimolares de histonas, excepto H1.
- Micrografías electrónicas de la cromatina revelaron que consiste en partículas de aproximadamente 100 Å de diámetro conectadas mediante ADN desnudo (como un collar de cuentas) y son aparentemente las responsables del patrón de rayos X.
- Controlando el tiempo de digestión de la cromatina con la nucleasa de micrococo (que rompe la doble hebra del ADN), se obuvieron fragmentos cuyo ADN tenía tamaños siempre múltiplos de, aproximadamente, 200 pares de bases (cuantificado mediante experimentos de electroforesis en gel).
- Experimentos de entrecruzamiento indicaron que las histonas H3 y H4 se asocian para formar el heterotetrámero (H3)2(H4)2.
Las observaciones de Kornberg lo llevaron a concluir que el nucleosoma está formado por el octámero (H2A)2(H2B)2(H3)2(H4)2, además de aproximadamente 200 pares de bases de ADN. En contacto directo con el centro del octámero hay 140-147 pares de bases. Entre dos octámeros adyacentes, el ADN espaciador tiene una longitud de 20-100 pb.[6] [7] [8] Kornberg postuló que la quinta histona, H1, estaba asociada de alguna manera en el exterior del nucleosoma.[5]
Cuando el ADN se replica in vivo, las hebras hijas son incorporadas inmediatamente en nucleosomas. Cuando el ADN se replica en presencia de cicloheximida (inhibidor de la síntesis de proteínas), sólo una hebra hija tiene nucleosomas.
Posteriormente en 1987, los investigadores Lorch et al. demostraron in vitro el rol de los nucleosomas como reguladores de la transcripción[9] y en 1988, Hans y Grunstein[10] y Clark-Adams et al.[11] lo demostraron in vivo.
Estructura
Partícula central del nucleosoma
La partícula central del nucleosoma se compone de una región de ADN de 140-147 nucleótidos de longitud, la cual envuelve el octámero de histonas en 1,7 vueltas en un superenrollamiento negativo o levógiro. El octámero se compone de las histonas centrales H2A, H2B, H3 y H4, cada una en parejas de dos.[13] Entre cada nucleosoma consecutivo existe un fragmento de ADN, llamado ADN espaciador, cuya longitud varía entre 20 y 100 nucleótidos en función de la especies y el tipo celular. La estructura completa conforma una hélice de 11 nm de diámetro y 5,5 nm de altura.[14][15][16][17]
Estas partículas se pueden observar mediante microscopía electrónica cuando la cromatina se encuentra en interfase y se fuerza su desempaquetamiento mediante una digestión parcial con ADNasa. Esto genera una estructura con forma de "collar de cuentas o de perlas", donde la cuerda del collar se corresponde al ADN y las cuentas/perlas a los nucleosomas.[18] La digestión con ADNasa de las regiones de ADN más accesibles, aquellas que no están unidas al nucleosoma, genera fragmentos de longitudes múltiplo de, aproximadamente, 200 nucleótidos. Mediante la técnica de electroforesis en gel se puede observar este patrón de fragmentación. Este tipo de digestión del ADN también puede ocurrir en condiciones naturales durante la apoptosis o muerte celular programada.[19]
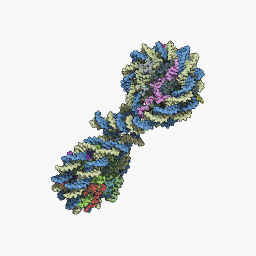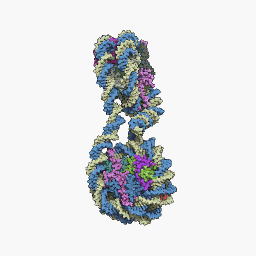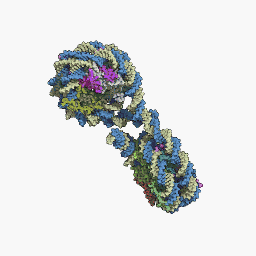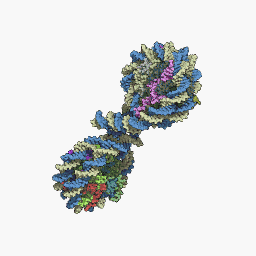
Los nucleosomas forman grupos unidos de ADN de modo que no se puede transcribir. Para que se produzca la transcripción, el ADN debe separarse de las histonas. En estado desplegado se conoce como: Eucromatina. Actualmente se cree que la eucromatina corresponde a la configuración de 11nm y la heterocromatina a la de 30nm.
Los genes residen en el ADN y contienen regiones llamadas exones que se traducen en proteínas, y otras denominadas intrones que están dispersas entre los exones y no se transcriben en proteínas.
El enrollamiento de la molécula de ADN en torno al nucleosoma reduce hasta en 6 veces la longitud de la cadena.
Referencias
- «Nucleosoma | NHGRI». Genome.gov. Consultado el 16 de abril de 2021.
- Viviana Sabbatino, Andrea Lassalle, Gladys Gálvez, Silvia Márquez (2011). «Niveles de organización de la Cromatina en: El Núcleo Celular». Guías temáticas: Biología celular y humana (Genomasur).
- White, Cindy L.; Suto, Robert K.; Luger, Karolin (17 de septiembre de 2001). «Structure of the yeast nucleosome core particle reveals fundamental changes in internucleosome interactions». The EMBO Journal 20 (18): 5207-5218. ISSN 0261-4189. PMC 125637. PMID 11566884. doi:10.1093/emboj/20.18.5207. Consultado el 20 de septiembre de 2022.
- Olins, Ada L.; Olins, Donald E. (25 de enero de 1974). «Spheroid Chromatin Units (ν Bodies)». Science (en inglés) 183 (4122): 330-332. ISSN 0036-8075. doi:10.1126/science.183.4122.330. Consultado el 20 de septiembre de 2022.
- Kornberg, Roger D. (24 de mayo de 1974). «Chromatin Structure: A Repeating Unit of Histones and DNA: Chromatin structure is based on a repeating unit of eight histone molecules and about 200 DNA base pairs.». Science (en inglés) 184 (4139): 868-871. ISSN 0036-8075. doi:10.1126/science.184.4139.868. Consultado el 20 de septiembre de 2022.
- Del Castillo Ruiz V, Uranga Hernández RD, Zafra de la RosaG (2012). Genética Clínica. México.: El manual moderno.
- Herráez A (2012). Biología Molecular e Ingeniería Genética (2ª. edición). España.: Elsevier.
- Garrido A, Teijón JM (2006). Fundamentos de bioquímica estructural (segunda edición). Madrid: Tebar,.
- Lorch, Yahli; LaPointe, Janice W.; Kornberg, Roger D. (24 de abril de 1987). «Nucleosomes inhibit the initiation of transcription but allow chain elongation with the displacement of histones». Cell (en english) 49 (2): 203-210. ISSN 0092-8674. PMID 3568125. doi:10.1016/0092-8674(87)90561-7. Consultado el 20 de septiembre de 2022.
- Han, Min; Grunstein, Michael (23 de diciembre de 1988). «Nucleosome loss activates yeast downstream promoters in vivo». Cell (en english) 55 (6): 1137-1145. ISSN 0092-8674. PMID 2849508. doi:10.1016/0092-8674(88)90258-9. Consultado el 20 de septiembre de 2022.
- Clark-Adams, C. D.; Norris, D.; Osley, M. A.; Fassler, J. S.; Winston, F. (1 de febrero de 1988). «Changes in histone gene dosage alter transcription in yeast.». Genes & Development (en inglés) 2 (2): 150-159. ISSN 0890-9369. PMID 2834270. doi:10.1101/gad.2.2.150. Consultado el 20 de septiembre de 2022.
- Harp, J. M.; Hanson, B. L.; Timm, D. E.; Bunick, G. J. (2000-12). «Asymmetries in the nucleosome core particle at 2.5 A resolution». Acta Crystallographica. Section D, Biological Crystallography 56 (Pt 12): 1513-1534. ISSN 0907-4449. PMID 11092917. doi:10.1107/s0907444900011847. Consultado el 20 de septiembre de 2022.
- Luger, Karolin; Mäder, Armin W.; Richmond, Robin K.; Sargent, David F.; Richmond, Timothy J. (1997-09). «Crystal structure of the nucleosome core particle at 2.8 Å resolution». Nature (en inglés) 389 (6648): 251-260. ISSN 1476-4687. doi:10.1038/38444. Consultado el 20 de septiembre de 2022.
- Harp, J. M.; Uberbacher, E. C.; Roberson, A. E.; Palmer, E. L.; Gewiess, A.; Bunick, G. J. (1 de marzo de 1996). «X-ray Diffraction Analysis of Crystals Containing Twofold Symmetric Nucleosome Core Particles». Acta Crystallographica Section D: Biological Crystallography (en inglés) 52 (2): 283-288. ISSN 0907-4449. doi:10.1107/S0907444995009139. Consultado el 20 de septiembre de 2022.
- Felsenfeld, Gary; Groudine, Mark (2003-01). «Controlling the double helix». Nature (en inglés) 421 (6921): 448-453. ISSN 1476-4687. doi:10.1038/nature01411. Consultado el 20 de septiembre de 2022.
- Hanson, B. Leif; Alexander, Chad; Harp, Joel M; Bunick, Gerard J (1 de enero de 2003). Preparation and Crystallization of Nucleosome Core Particle. Chromatin and Chromatin Remodeling Enzymes, Part A (en inglés) 375. Academic Press. pp. 44-62. doi:10.1016/s0076-6879(03)75003-4. Consultado el 20 de septiembre de 2022.
- Alberts, Bruce; Wilson, John; Hunt, Tim (2008). Molecular biology of the cell (5th ed edición). Garland Science. ISBN 978-0-8153-4105-5. OCLC 82473851. Consultado el 20 de septiembre de 2022.
- Alberts, B. (2009). Essential Cell Biology (2nd edición). New York: Garland Science.
- Stryer, Lubert (1995). Biochemistry (4th ed edición). W.H. Freeman. ISBN 0-7167-2009-4. OCLC 30893133. Consultado el 20 de septiembre de 2022.
Enlaces externos
- Ilustración de la estructura del nucleosoma mediante modelos moleculares tridimensionales interactivos empleando Java (enlace roto disponible en Internet Archive; véase el historial, la primera versión y la última). o empleando MDL Chime
- https://web.archive.org/web/20140903092913/http://medmol.es/glosario/23/
